
Un nuovo studio porta alla luce la diversità umana.
Di recente è stata pubblicata la prima bozza del pangenoma umano, che cattura la diversità umana tramite il DNA di diverse persone. Indice dei contenuti…



Di recente è stata pubblicata la prima bozza del pangenoma umano, che cattura la diversità umana tramite il DNA di diverse persone. Indice dei contenuti…
I batteri sono microrganismi unicellulari, aploidi, in grado di riprodursi autonomamente nell’ambiente e anche in vari tessuti del corpo umano; vengono utilizzati per questo nei laboratori….

Le 5 basi azotate del DNA e dell’RNA, sono state ritrovate in frammenti di meteoriti. Gli scienziati, in passato, erano riusciti a trovare solo tre…
Dopo due decenni, i ricercatori hanno generato la prima sequenza completa e senza interruzioni di un genoma umano. Indice articolo: Cos’è un genoma?…

Il Nobel per la Chimica quest’anno è stato “vinto a parimerito” dalla chimica americana Jennifer A. Doudna e dalla biochimica francese Emmanuelle Charpentier. Il metodo di modificazione del DNA…

l Nobel per la Chimica quest’anno è stato “vinto a parimerito” dalla chimica americana Jennifer A. Doudna e dalla biochimica francese Emmanuelle Charpentier, per lo sviluppo di CRISPR Cas9, una tecnica di editing…
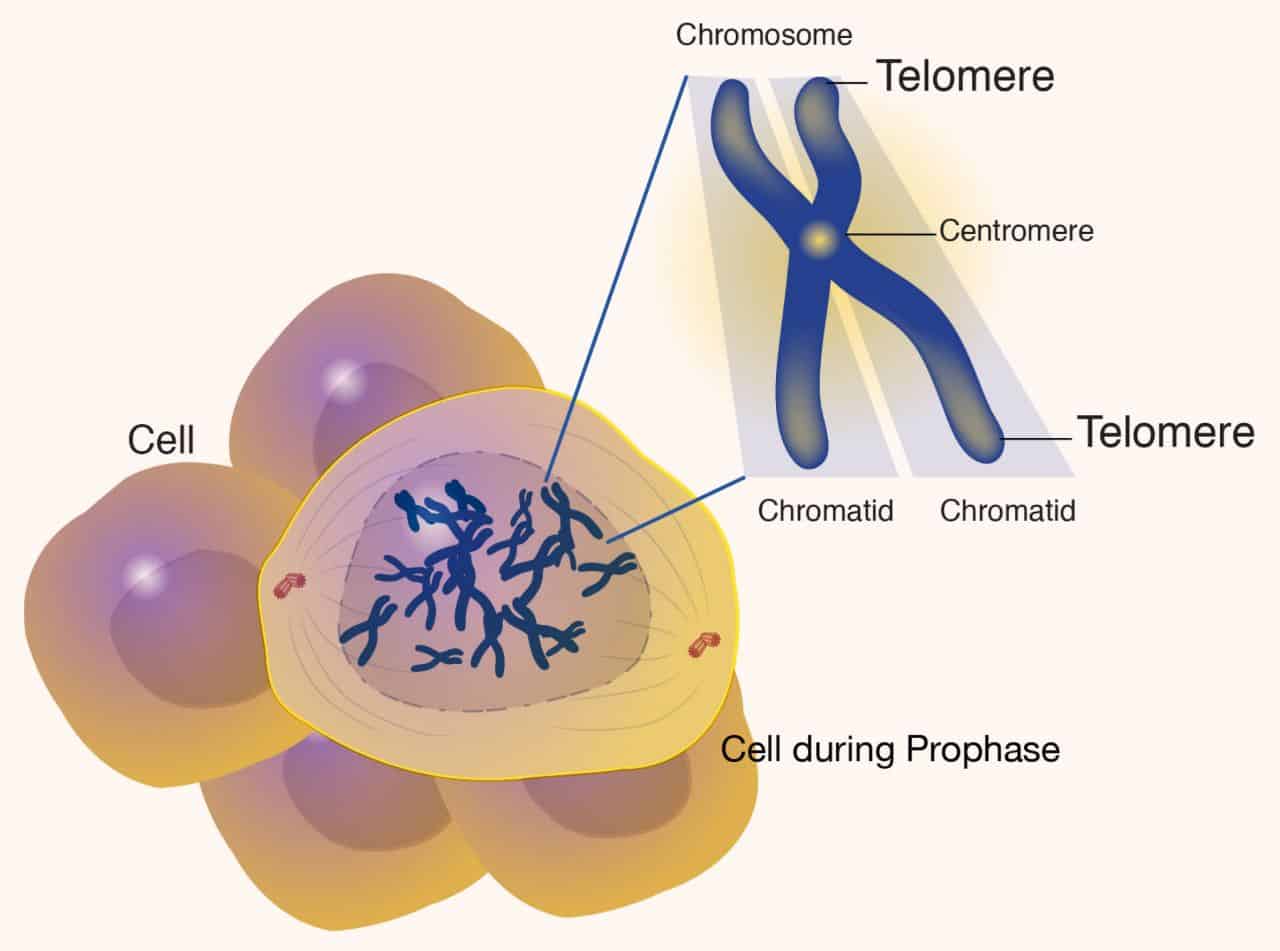
Progresso tecnologico e speranze future per le patologie incurabili. Un team di scienziati statunitensi e britannici ha prodotto la sequenza di DNA senza interruzioni, dall’inizio…
Dopo aver passato la prima parte di 2020 quasi totalmente in lockdown, stiamo imparando a convivere con il virus e contemporaneamente stiamo conoscendo la sua…

L’estate è ormai iniziata da molto, almeno secondo il calendario, la sessione estiva è terminata da poco e, che sia andata bene o male, ormai…